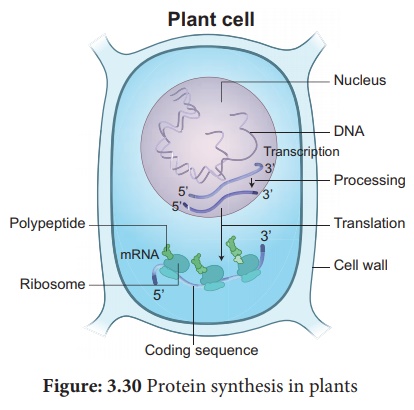
Le processus de synthèse des protéines comprend deux étapes principales, la transcription et la traduction.
1. Transcription
La transcription est le processus dans lequel un brin d'ADN agit comme une matrice pour générer de l'ARNm avec les bases complémentaires du brin matrice. Elle est catalysée par des enzymes appelées ARN polymérases.
La transcription et le traitement de l'ARN ont lieu dans le noyau , tandis que la traduction se produit dans les ribosomes trouvés dans le cytoplasme . Chez les eucaryotes, les molécules d'ARNm sont monocistroniques , une seule protéine étant dérivée de chaque ARNm.
La transcription commence par le déroulement de la double hélice d'ADN et les liaisons hydrogène sont rompues au site du gène en cours de transcription.
Brin modèle / Brin non codant / Brin antisens
Le brin d'ADN orienté dans la direction 3 ' → 5' qui sert de matrice pour la synthèse de l'ARNm est appelé brin matrice.
Volet de codage / Volet sans modèle / Volet de détection
L'autre brin d'ADN qui n'est pas transcrit est appelé le brin codant.
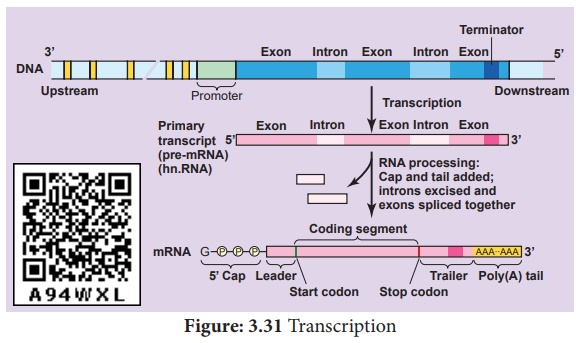
Une séquence spécifique de nucléotides d'ADN appelée promoteur est nécessaire pour que la transcription ait lieu. Il se compose d'une boîte TATA et d'un site de début de transcription où la transcription commence.
Les séquences de terminaison sont les séquences d'ADN qui indiquent quand l'ARN polymérase doit cesser de produire une molécule d'ARN.
Le gène de structure eucaryote a 3 caractéristiques dans le promoteur
1. Éléments réglementaires
2. Boîte TATA
3. Un site de départ transcriptionnel
Le site d'initiation de la transcription contient environ 25 pb (paires de bases) en amont, la séquence est TATAAT connue sous le nom de TATA ou Hogness box qui est présente dans le promoteur central. Les facteurs transcriptionnels généraux sont les protéines qui reconnaissent les séquences de bases de l'ADN et contrôlent la transcription. Certains facteurs de transcription se lient directement au promoteur.
Certains facteurs de transcription reconnaissent les éléments régulateurs et s'y lient pour augmenter le taux de transcription, d'autres inhibent la transcription.
Pour démarrer le processus de transcription, les éléments régulateurs aident l'ARN polymérase à reconnaître le promoteur principal. Les deux catégories d'éléments réglementaires sont
1. Séquences amplificatrices - ce sont des séquences d'ADN (séquences d'activation) qui aident à influencer la transcription.
2. Séquence silencieuse - séquences d'ADN qui inhibent ou diminuent la transcription.
Séquence consensus - Une séquence idéale dans laquelle chaque position représente la base qui se trouve le plus souvent.
En plus des facteurs de transcription généraux (GTF) et de l'ARN Pol II, un médiateur est requis pour la transcription. Les interactions entre l'ARN polymérase II et le TF régulateur qui se lient aux activateurs ou aux silencieux sont médiées par un médiateur.
Les ARN polymérases ne peuvent pas se lier directement à l'ADN, elles se lient d'abord au facteur de transcription qui reconnaît les séquences promotrices qui aident à trouver les régions codant pour les protéines de l'ADN.
L'ARN polymérase avec la séquence promotrice transcrira le gène. Le facteur de transcription joue un rôle important dans le guidage des ARN polymérases vers la séquence promotrice. Les ARN polymérases lient ensemble les nucléotides d'ARN en formant un brin en croissance dans la direction 5 ' → 3'. La transcription se produit dans la direction 5 ' → 3', l'ARN polymérase catalyse l'addition de nucléotides à l'extrémité 3' de la chaîne en croissance de l'ARN.
Chez les eucaryotes, on trouve 3 ARN polymérases différentes appelées ARN polymérases I, II et III.

Le traitement du pré-ARNm en ARNm mature / Mécanisme moléculaire de la modification de l'ARN
Chez les eucaryotes, trois types principaux d'ARN, l'ARNm, l'ARNt et l'ARNr sont produits à partir d'une molécule d'ARN précurseur appelée transcrit primaire ou préARN. L'ARN polymérase II transcrit le précurseur de l'ARNm, également appelé ARN nucléaire hétérogène ou hnARN, qui est traité dans le noyau avant d'être transporté dans le cytoplasme.
Plafonnement
La modification à l'extrémité 5' du transcrit d'ARN primaire (ARN hn) avec du triphosphate de méthylguanosine est appelée coiffage.
Objectif du plafonnement
1. Protège l'ARN de la dégradation.
2. Le coiffage joue un rôle important dans l'élimination du premier intron dans le pré-ARNm.
3. Il régule l'exportation d'ARNm du noyau vers le cytoplasme.
4. Il aide à la liaison de l'ARNm au ribosome.
Tailing / Polyadénylation
L'extrémité 3 'de l'ARNhn est clivée par une endonucléase et une chaîne de nucléotides d'adénine est ajoutée à l'extrémité 3' de l'ARNhn (pré ARNm) est connue sous le nom de queue Poly (A) - Polyadénylation. Ce processus est appelé queue ou polyadénylation.
But du suivi
1. La traduction du transcrit d'ARN est facilitée.
2. Aide à la synthèse des Polypeptides.
3. Il améliore la stabilité de l'ARNm dans le cytoplasme.
Les régions codant pour les protéines ne sont pas continues chez les eucaryotes. Les gènes fractionnés ont été découverts indépendamment par Richard J Roberts et Phillip A. Sharp en 1977 et ont reçu le prix Nobel en 1993. Les exons sont les séquences codantes ou les séquences exprimées contiennent des informations biologiques dans l'ARNm traité mûri. Les introns sont des séquences intermédiaires, qui sont des séquences non codantes (séquences non codantes d'acides aminés) qui doivent être retirées d'un gène avant la fabrication du produit d'ARNm. Les introns ne codent pour aucune enzyme ou protéine structurale ou polypeptides. Ces exons et introns sont connus sous le nom de Split Genes.
2. Épissage d'ARN chez les plantes
L'épissage d'ARN est un processus qui implique la découpe ou l'élimination des introns et le tricotage des exons. Ce processus se déroule dans des particules sphériques qui sont un complexe multiprotéique appelé SPLICISOMES. Il mesure environ 40 à 60 nm de diamètre. Les spliceosomes ont de nombreux petits acides ribonucléiques nucléaires (ARNsn) et de petites particules de protéines ribonucléaires nucléaires (snRNP) qui identifient et aident à éliminer les introns.
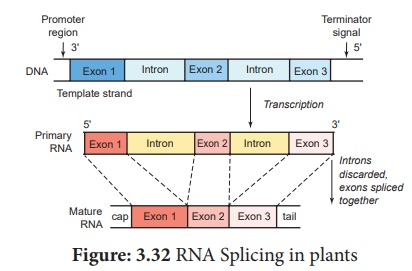
Un spliceosome élimine les introns avec un ribozyme enzymatique. Maintenant, l'ARNm mature se détache des spliceosomes par le pore nucléaire et est transporté du noyau dans le cytoplasme, et s'attache au ribosome pour effectuer la traduction. L'ARN et de nombreuses protéines sont transportés à travers un complexe de pores nucléaires par un processus dépendant de l'énergie.
3. Traduction
L'information génétique dans le code ADN est copiée sur l'ARNm lié dans les ribosomes pour fabriquer des polypeptides. La séquence nucléotidique de l'ARNm est décodée en séquence d'acides aminés de la protéine qui est catalysée par le ribosome. Ce processus s'appelle la traduction.
Terminologie dans la synthèse des protéines
Codon - Les codes ADN sont appelés codes triplet et ceux de l'ARNm sont appelés codons. Chaque triplet spécifie un acide aminé particulier. Les codons présents dans l'ARNm sont lus dans la direction 5' → 3'. Il existe 64 codons dont 61 codons pour les acides aminés.
Codon de démarrage - méthionine spécifiée par AUG
Codon d'arrêt ou de terminaison - UAA - Ocre UAG - ambre et UGA - Opale.
Anticodons - Le triplet de bases dans une molécule d'ARNt est appelé anticodon. Dans l'ARNt, la séquence de trois bases complémentaires des codons de l'ARNm est appelée anticodon. Les codons de l'ARNm sont reconnus par les anticodons de l'ARNt qui sont orientés dans la direction 3' → 5'.
Processus de traduction
Voici les principales étapes du processus de traduction
1. Initiation
La traduction commence par le codon AUG (start codon) de l'ARNm. La traduction se produit à la surface de l'arène macromoléculaire appelée ribosome. C'est un organite non membraneux. Au cours du processus de traduction, les deux sous-unités de ribosomes s'unissent (se combinent) et maintiennent l'ARNm entre elles. La synthèse protéique commence par la lecture des codons de l'ARNm. L'ARNt apporte l'acide aminé au ribosome, une machine moléculaire qui unit les acides aminés en une chaîne selon les informations données par l'ARNm. L'ARNr joue le rôle structurel et catalytique lors de la traduction.
Un ribosome a un site de liaison pour l'ARNm et deux pour l'ARNt. Les deux sites de liaison de l'ARNt sont
je. P-Site - Le site de liaison peptidyl - ARNt est l'un des sites de liaison de l'ARNt. Sur ce site, l'ARNt est retenu et lié à l'extrémité croissante de la chaîne polypeptidique.
ii. A-Site - Le site de liaison Aminoacyl - ARNt . Il s'agit d'un autre site de liaison à l'ARNt qui contient les acides aminés entrants appelés aminoacyl ARNt. Les anticodons de l'ARNt s'apparient avec les codons de l'ARNm dans ces sites.
2. Allongement de la chaîne polypeptidique
Les sites P et A sont proches, de sorte que deux ARNt forment des paires de bases avec un codon adjacent. La chaîne polypeptidique est formée par l'appariement de codons et d'anticodons selon la séquence nucléotidique de l'ARNm.
Traducteurs du code génétique – ARNt
L'ARNt traduit le code génétique de la séquence d'acide nucléique à la séquence d'acides aminés, c'est-à-dire du gène - Polypeptide. Lorsqu'un acide aminé est attaché à l'ARNt, il est appelé aminoacylé ou chargé. Il s'agit d'un processus nécessitant de l'énergie qui
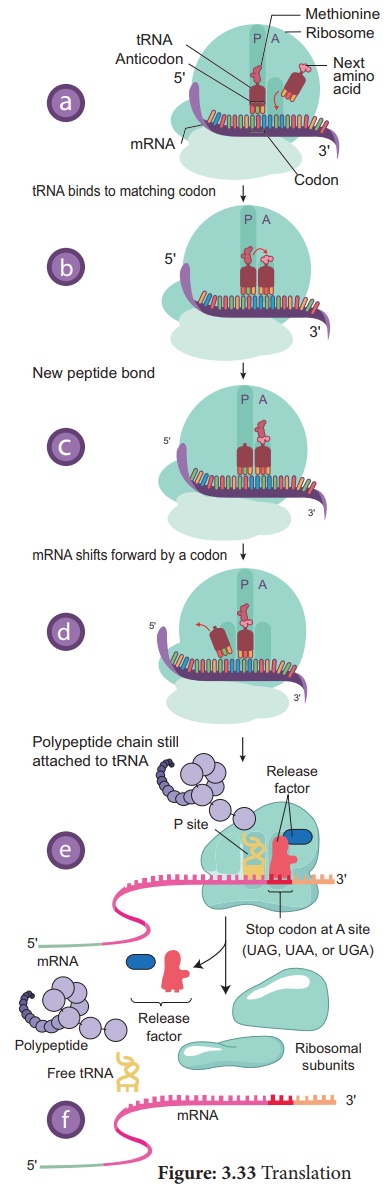
La traduction commence par le codon AUG (start codon) de l'ARNm. L'ARNt qui porte le premier acide aminé méthionine se fixe au site P du ribosome. Le ribosome ajoute de nouveaux acides aminés aux polypeptides en croissance. Les deuxièmes molécules d'ARNt ont des anticodons qui portent des paires d' alanine d'acides aminés avec le codon d'ARNm dans le site A du ribosome. Les acides aminés méthionine et alanine sont suffisamment proches pour qu'une liaison peptidique se forme entre eux.
La liaison entre le premier ARNt et la méthionine se rompt maintenant. Le premier ARNt quitte le ribosome et le site P est vacant. Le ribosome déplace maintenant un codon le long du brin d'ARNm. La deuxième molécule d'ARN-t occupe maintenant le site P. Le troisième t-ARN vient remplir le site A (sérine). Maintenant, une liaison peptidique est formée entre l' alanine et la sérine. L'ARNm se déplace ensuite à travers le ribosome par trois bases. Cela expulse l'ARNt désacylé / non chargé du site P et déplace l'ARNt peptidyle dans le site P et vide le site A. Ce mouvement d'ARNt du site A au site P est appelé translocation. La translocation nécessite l'hydrolyse du GTP.
Le ribosome (ribozyme - peptidyl transférase) catalyse la formation d' une liaison peptidique en ajoutant un acide aminé à la chaîne polypeptidique en croissance.
Le ribosome se déplace de codon en codon le long de l'ARNm dans le sens 5' vers 3'. Les acides aminés sont ajoutés un par un traduits en polypeptide comme dicté par l'ARNm. La traduction est un processus énergivore. Un groupe de ribosomes sont liés entre eux par une molécule d'ARNm et formant le site de synthèse des protéines est appelé polysomes ou polyribosomes.
3. Arrêt de la synthèse polypeptidique
Les eucaryotes ont des protéines cytosoliques appelées facteurs de libération qui reconnaissent le codon de terminaison, UAA, UAG ou UGA lorsqu'il se trouve dans le site A. Lorsque le ribosome atteint un codon stop, la synthèse protéique s'arrête. Les ribosomes sont donc les usines de fabrication de protéines d'une cellule. Lorsque le polypeptide est terminé, le ribosome libère le polypeptide et se détache de la molécule d'ARNm. Maintenant, le ribosome se divise en petites et grandes sous-unités après la libération de l'ARNm.
4. Épissage alternatif dans les usines
Il est très utile dans la régulation de l'expression des gènes pour surmonter le stress environnemental chez les plantes.
L'épissage alternatif est un mécanisme/processus important par lequel plusieurs produits d'ARNm et de protéines multiples peuvent être générés à partir d'un seul gène. Les différentes protéines générées sont appelées isoformes. Il existe différents modes d'épissage alternatif.
Lorsque plusieurs introns sont présents dans un gène, ils sont éliminés séparément ou en tant qu'unité. Dans certains cas, un ou plusieurs exons présents entre les introns sont également éliminés.
Importance de l'épissage alternatif
1. Les protéines transcrites à partir d'ARNm à épissage alternatif contenant différentes séquences d' acides aminés conduisent à la génération de diversité protéique et de fonctions biologiques.
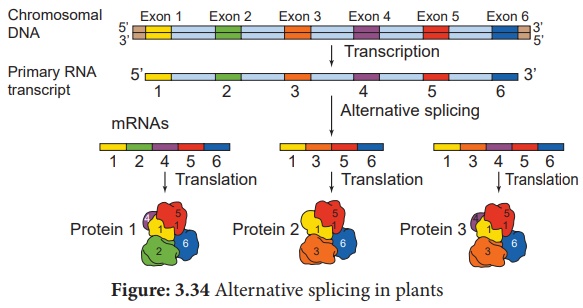
2. Plusieurs isoformes de protéines sont formées.
3. Il crée plusieurs transcrits d'ARNm à partir d'un seul gène. L'invention concerne un processus de production de protéines apparentées à partir d'un seul gène permettant ainsi d'augmenter le nombre de produits géniques.
4. Il joue un rôle important dans les fonctions des plantes telles que la réponse au stress et la sélection des traits.
La plante s'adapte ou se régule à l'évolution de l'environnement.
5. Édition d'ARN - Traitement post-transcriptionnel de l'ARN dans les plantes
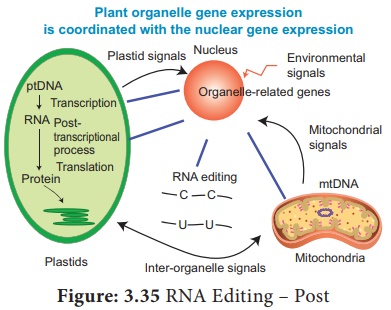
Figure : 3.35 Édition de l'ARN - Post
Traitement transcriptionnel de l'ARN chez les plantes La modification chimique telle que la modification de base, l'insertion ou la délétion de nucléotides et le remplacement de nucléotides de l'ARNm entraîne l'altération de la séquence d'acides aminés de la protéine qui est spécifiée est appelée édition de l'ARN.
Cela entraîne le changement de la séquence codant pour la protéine de l'ARN après la transcription.
Les propriétés de codage du transcrit d'ARN sont modifiées. L'information génétique codée dans le génome du chloroplaste est altérée par un phénomène post-transcriptionnel qui est spécifique au site (C → U) dans le chloroplaste des plantes supérieures - l'édition de l'ARN se produit dans les mitochondries et le chloroplaste des plantes.
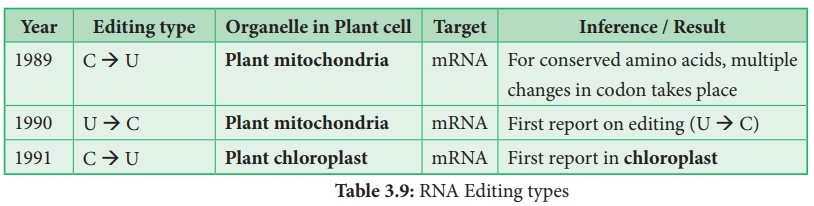
Dans les cellules végétales, l'édition de l'ARN par des transitions de pyrimidine se produit dans les mitochondries et les plastes (chloroplastes). Il existe deux principaux types d'édition d'ARN. (1) Édition par substitution - Modification de bases nucléotidiques individuelles. ARN des mitochondries et des chloroplastes chez les plantes. (2) Modification par insertion / suppression - Les nucléotides sont ajoutés ou supprimés du nombre total de bases.
Importance de l'édition d'ARN
1. Dans le chloroplaste végétal supérieur, il aide à restaurer les codons pour les acides aminés conservés, notamment le codon d'initiation et de terminaison.
2. Il régule l'expression des gènes Organellar dans les plantes.
3. L'édition de l'ARN entraîne la restauration des codons pour les résidus d'acides aminés conservés phylogénétiquement.
6. Gènes sauteurs

Avez-vous entendu parler de Jumping Genes ou de Hopping Genes ?
C'est le surnom des éléments génétiques transposables. Les transposons sont les séquences d'ADN qui peuvent se déplacer d'une position à une autre position dans un génome. Cela a été signalé pour la première fois en 1948 par la généticienne américaine Barbara McClintock comme «élément de contrôle mobile» dans le maïs. L'un des scientifiques les plus importants du 20ème siècle était Barbara McClintock parce qu'elle a donné un changement dans l'organisation des gènes. McClintock a reçu le prix Nobel en 1983 pour ses travaux sur les transposons. Barbara McClintock, lorsqu'elle a étudié l'aleurone de grains de maïs simples, a noté l'héritage instable du motif en mosaïque de taches bleues, brunes et rouges en raison de la production différentielle d'anthocyanes vacuolaires.
Dans le génome de la plante de maïs, le transposon AC / Ds (AC = Activator, Ds = Dissociation). L'activité de l'élément AC est très distincte dans la plante de maïs. La transposition dans les cellules somatiques entraîne des changements dans l'expression des gènes tels que la pigmentation panachée dans les grains de maïs. Le génome du maïs contient des éléments transposables qui régulent les différents motifs de couleur des grains.
Les découvertes de McClintock ont conclu que les gènes Ds et AC étaient des éléments de contrôle mobiles. Nous l'appelons maintenant comme éléments transposables, un terme inventé par le généticien du maïs, Alexander Brink. McClintock a donné la première preuve expérimentale directe que les génomes ne sont pas statiques mais sont des entités hautement plastiques.
Signification des transposons
1. Ils contribuent à de nombreuses mutations visibles et à un taux de mutation dans un organisme.
2. Au cours de l'évolution, ils contribuent à la diversité génétique.
3. Dans la recherche génétique, les transposons sont des outils précieux qui sont utilisés comme mutagènes, comme marqueurs de clonage, comme véhicules pour insérer de l'ADN étranger dans un organisme modèle.
Génome végétal - Le mot génome est défini comme l'ensemble complet de l'ADN (y compris tous les gènes et les régions intergéniques) présent dans un organisme. Il spécifie l'ensemble des informations biologiques d'un organisme. Il existe trois génomes distincts dans les cellules eucaryotes et ils sont (1) Le génome nucléaire (2) Le génome mitochondrial et (3) Le génome chloroplastique présent uniquement dans les plantes.
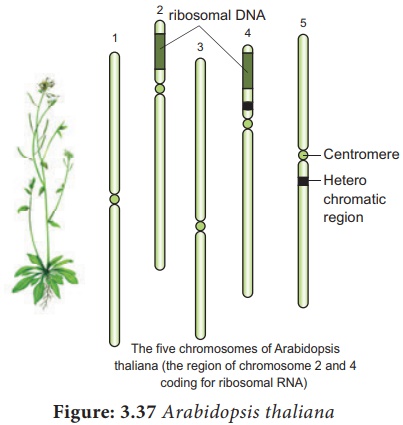
Arabidopsis thaliana - Thale cresson, cresson de souris
1. C'est une plante modèle pour l'étude des aspects génétiques et moléculaires du développement des plantes.
2. Il appartient à la famille de la moutarde et c'est la première plante à fleurs, où tout son génome est séquencé.
3. Les deux régions de l'ADN ribosomal organisateur nucléolaire qui codent pour l'ARN ribosomal sont présentes à l'extrémité des chromosomes 2 et 4
4. C'est une plante diploïde ayant un petit génome avec 2n = 10 chromosomes. Plusieurs générations peuvent être produites en une année. Cela facilite donc l'analyse génétique rapide. Le génome a un ADN peu répétitif, plus de 60% de l'ADN nucléaire ont des fonctions de codage des protéines.
5. La plante est petite, autofécondée, plante annuelle à long jour avec un cycle de vie court (seulement 6 semaines), un grand nombre de graines sont produites et elles sont faciles à cultiver en laboratoire. Il est facile d'induire des mutations. Il possède de nombreuses ressources génomiques et la transformation peut se faire facilement.
6. En 1982, Arabidopsis a achevé avec succès son cycle de vie en microgravité, c'est-à-dire dans l'espace. Cela montre que les missions spatiales humaines avec des compagnons végétaux peuvent être possibles.

